对于公共测序数据的分析,好多二代测序的数据都储存在GEO数据库以及 SRA 这样的平台。之前介绍过的ARCHS4-公共二代测序数据下载数据库就是把 GEO 的很多 [RNA-seq的数据进行了统一重新分析最后组合成一个大型数据集。除了 ARCHS4 之外还有其他的大型数据集比如今天要介绍的这个:Digital Expression Explorer 2(DEE2): http://dee2.io/index.html
背景数据集介绍
DDE2 收集了GEO/SRA的RNA-seq 数据。在收集相关数据之后,利用统一分析流程进行处理。
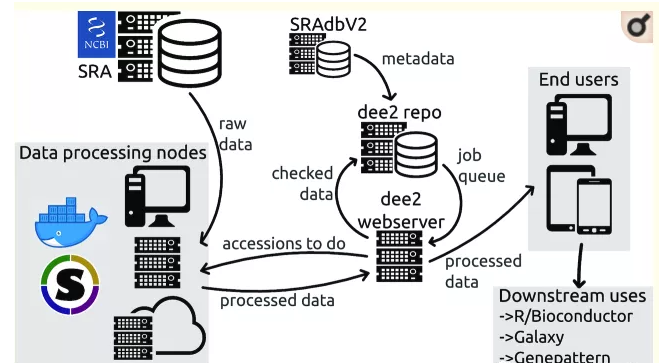
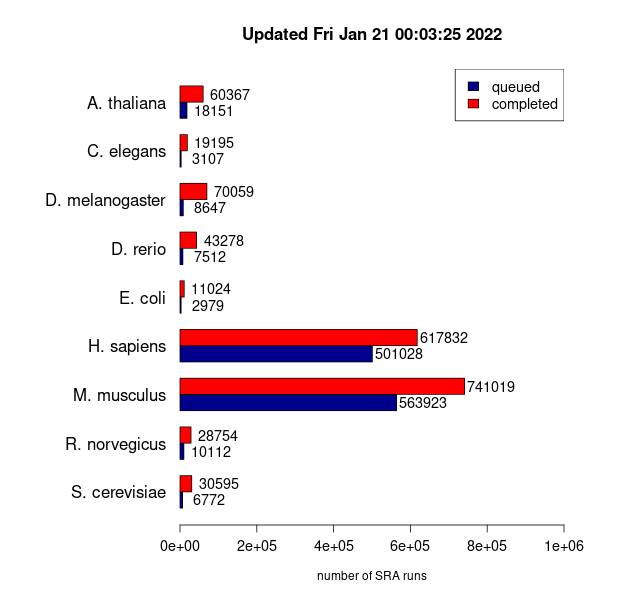
经过收集和分析。最终得到了多个物种的RNA-seq Count数据。其中目前人类当中就包括 617832 个测序数据样本。
数据库使用
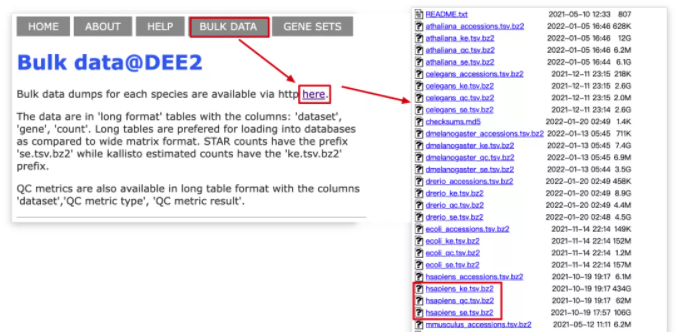
作为一个储存大量测序数据集的平台,主要的功能就是下载经过处理的 RNA-seq 的数据。我们可以点击Bulk DATA 直接下载分析的所有数据
同时也可以在下面选择感兴趣的物种,然后输入关键词/GEO ID 检索。例如搜索:“GSE63462”
这里选择物种是必须的,不然就算输入 GSE /关键词都检索不到结果
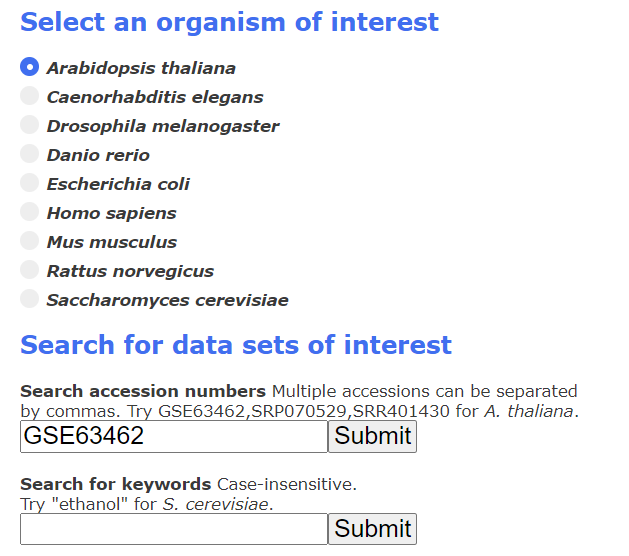
输入之后就得到检索结果了。我们可以选择想要下载的内容。点击Get Counts 就可以下载到这些测序数据的Count数据了。
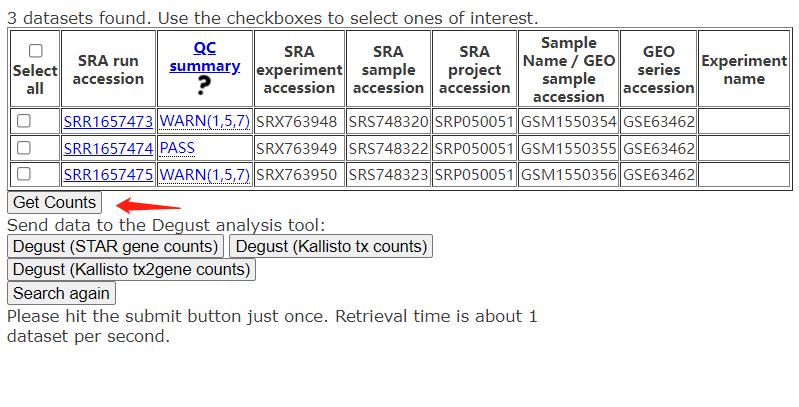
下载之后是一个压缩包。压缩包里面有一个README. md文件。用文本文档打开就可以知道具体每一个文件是什么内容了
同时如果想要进行一些基本的差异分析的话,也可以直接把 Count 数据发送到Degust进行基本的差异表达分析。
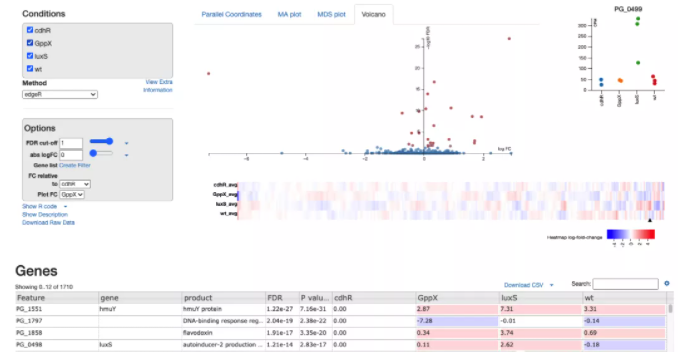
总的来说
以上就是 DEE2 的基本内容了。主要还是用来下载 RNA-seq 经过处理后的 Count 数据。一般来说测序数据从 Faseq 到 Count 需要很大的计算资源的。如果能得到 Count 数据。后续的就很容易分析了。比如想要做差异表达分析就可以直接把数据上传到:DEApp-差异表达分析工具进行分析即可。














